
Środowisko do realizacji projektów w architekturze aplikacji internetowej
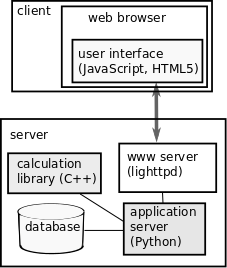
Środowisko wykorzystuje Python/C++/JavaScript oraz szereg bibliotek. Dostarcza trójwarstwowej architektury z warstwą danych, przetwarzania i prezentacji.
Wykorzystuje model aplikacji internetowej, warstwa trwałości oraz przetwarzania jest umieszczona na serwerze, warstwa prezentacji oraz częściowo przetwarzania
(badanie poprawności wprowadzonych danych, formatowanie wyglądu wyników) jest umieszczona po stronie klienta. Podstawowe moduły pokazano na rysunku.
Start
Aby zbudować prosty przykład aplikacji typu 'Hello World' proszę
pobierz aktualną wersję,
rozpakuj archiwum, zainstaluj narzędzia, tak jak opisano w pliku README_EN (plik ten jest umieszczony w głównym katalogu),
następnie uruchom komendę scons w katalogu, gdzie rozpakowałeś archiwum.
Aby uruchomić aplikację (klienta i serwer) lokalnie użyj komendy scons r=l.
Niekompletna i nieprzetestowana instrukcja instalacji środowiska pod Windows (zawiera zrzuty ekranów i realizuje kilka punktów opisanych w pliku README_EN
jest dostępna tutaj.
Zasoby
Jak cytować BioWeb?
Robert M. Nowak, Polyglot programming the applications to analyze genetic data, BioMed Research International, vol. 2014,
doi:10.1155/2014/253013, http://www.hindawi.com/journals/bmri/2014/253013
Przykładowe projekty
Przykładowa aplikacja jest opisana w sekcji Start. Dodatkowe, proste przykłady są wymienione niżej.
- Autor: Józef Marek.
Kodowanie i dekodowanie szyfrem Cezara. C++/Django/AngularJS.
CeasarCipher.zip
- Autor: Rafał Pilarczyk.
Filtrowanie obrazów filtrami 3x3, progowanie, zwiększanie kontrastu i jasności. C++/Django/AngularJS.
ImageFiltering.zip
- Autor: Krzysztof Lis.
Fazy księżyca w 2014 r. C++/Django/AngularJS.
DjangoMoon.zip
- Autor: Tomasz Iwaszko.
Wyznacznik macierzy kwadratowej dowolnego rozmiaru. C++/Web2py/JQuery.
MatrixDeterminant.zip
- Autor: Jakub Tomaszek.
Konwerter z/na leet speak. C++/Web2py/JQuery.
leet.zip
- Autor: Adam Stelmaszczyk. Prosty konwerter, który zamienia normalne obrazki w
ASCII-grafikę. Format obrazka dowolny, wskazujemy go przez podanie URL
do niego.
W Pythonie wczytuję obrazek o podanym URL i konwertuję go do skali
szarości, po czym wołam funkcję C++ przekazując listę wartości
jasności wszystkich pikseli.
W C++ przechodząc przez wszystkie piksele obrazka, tworzę ciąg znaków
ASCII, który potem wyświetlam. Kolor czarny odwzorowuje w "piksel
znakowy", który jest jak najciemniejszy, np. '#'. Troszkę jaśniejszy
kolor w '$', potem ciemny szary w '@' itd. Biały - w spacje.
ImageToAscii.zip.
C++/Web2py/Flex.
- Autor: Jakub Barejka. Jest to prosty kalkulator układów pokerowych.
Z talii kart poprzez odpowiednie przyciski losuję dwie karty gracza, a następnie pięć kart
stołu. Wtedy możliwe jest policzenie poprzez funkcję C++ najlepszego 5-kartowego układu (spośród podanych siedmiu kart). W efekcie wyświetlony
zostaje najlepszy możliwy układ jaki można z tych kart złożyć.
PokerVariants.zip.
C++/Web2py/Flex.
- Autor: Michał Uziak.
Skrypt dzieli wpisany tekst na wyrazy i liczy częstość ich wystąpienia. W
pierwsze pole tekstowe wkleja się tekst (bez znaków diakrytycznych). Po
kliknięciu przycisku "Count words" w dolnym polu tekstowym pojawi się
lista wyrazów, wraz z częstością wystąpień.
wordcounter.zip.
C++/Web2py/Flex.
- Autor: Michał Artur Krawczak.
Aplikacja, która oblicza datę Wielkanocy w zadanym roku, używa JavaScript z biblioteką jQuery do prezentacji wyników.
easter.zip.
C++/Web2py/Flex.
- Autor: Paweł Barszcz.
Program do rozwiązywania krzyżówek sudoku.
SudokuSolver.zip.
C++/Web2py/Flex.
- Autor: Bartosz Balcerek, Marcin Król.
Program do znajdowania największego wspólnego podzielnika gcd.zip
i największej wspólnej wielokrotności lcm.zip. C++/Web2py/Flex.
- Autor: Mariusz Cieśluk. Obliczanie rat kredytu. C++/Django/JQuery
CreditCalculation.zip
- Autor: Damian Kwiatosz. Generator Labiryntu. C++/web2py/JQuery
MazeGenerator.zip
- Autor: Kacper Michajłow. Formatowanie kodu C/C++ z użyciem biblioteki astyle.sourceforge.net. C++/web2py/JQuery
CodeBeautifier.zip
Oprogramowanie wykorzystujące bioweb
DNASynth, aplikacja do syntezy sztucznych genów
(publikacja,
kod źródłowy)
CodonHMM, oblicza sekwencję DNA na podstwie sekwencji białka używając ukrytych modeli Markowa
(publikacja,
kod źródłowy)
WebOmicsViewer, przeglądarka genomu ogórka
(publikacja,
kod źródłowy)
PETconn, aplikacja do łączenia kontigów wykorzystująca sekwencje sparowanych końców
(publikacja,
kod źródłowy)
DNAMarkers, analiza mieszanin DNA
(publikacja,
kod źródłowy)
GenomeCmp, wyszukuje rearanżacje w genomach
(publikacja)
DnaAssembler, asembler wykorzystujący graf de Brujna
(publikacja,
kod źródłowy (tylko moduł obliczeniowy, C++>)



